品质至上,客户至上,您的满意就是我们的目标
技术文章
当前位置: 首页 > 技术文章
Plant Phenomics | 基于Vision Transformers估算种子混合物的成分和营养价值
发表时间:2023-11-22 10:55:58点击:446
来源:植物表型组学
分享:

在欧洲,当地牧场的人们通过种植谷物和豆类的种子混合物,用于以较低的生产成本生产均衡且富含能量和蛋白质的动物饲料。这种方法符合欧洲植物检疫产品使用规定的发展方向,同时也满足了消费者对有机产品的需求。然而,由于谷物和豆类成熟的时间不同,造成产量差距大,人们难以评估其收获后的营养价值。
2023年11月,Plant Phenomics 在线发表了法国University Montpellier等单位题为Estimating Compositions and Nutritional Values of Seed Mixes Based on Vision Transformers 的研究论文。
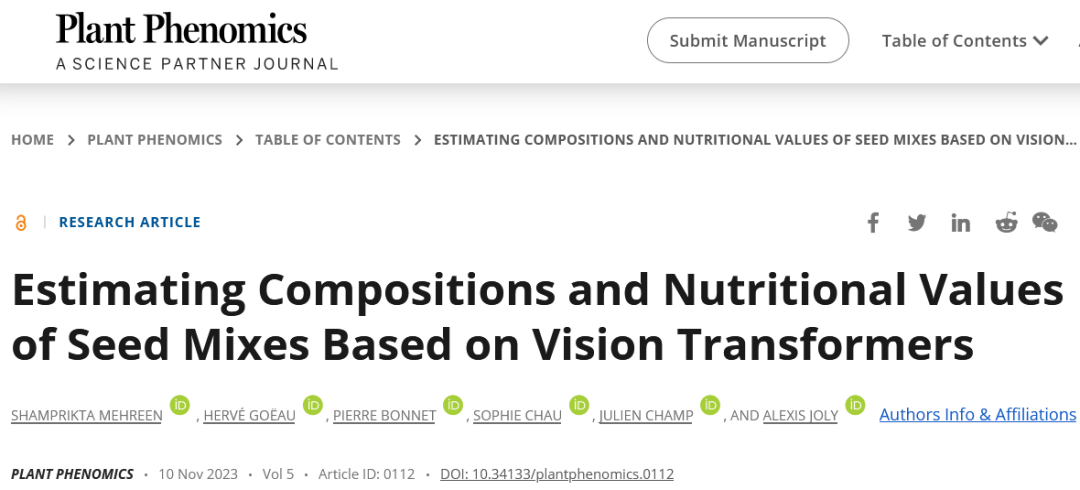
受Pl@ntNet的启发(一个参与式的公民科学平台,用于收集、共享和查找基于自动识别的植物观察结果),文章提出了一项新的技术:通过使用共建的种子混合物数据库,在EfficientNet和Vision Transformer两种最新的深度模型上进行训练,最后迁移到APP上自动估计收获种子混合物的营养价值,以帮助人们更好的管理产量。为此,本研究在2019年到2022年3年的时间里,从205份种子混合物中收集了约4749张图像(图1)。
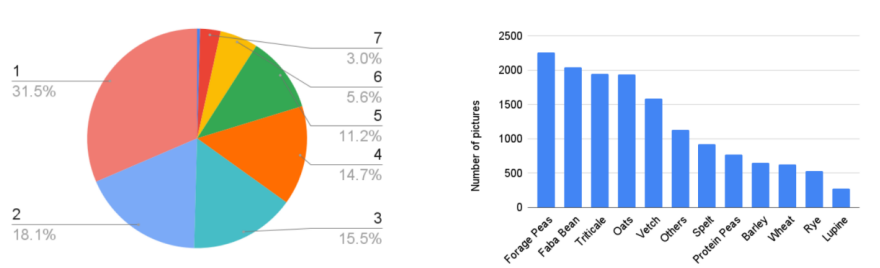
图1混合料中种子品种的分布情况以及每个品种的图片数量
本研究将种子混合物以8:2的比例随机划分为训练集和验证集,使用了多种数据增强技术,包括center crop, random rotation, color jitter, auto contrast和 RandAugment,在计算机视觉领域中两种主要深度学习模型CNN EfficientNet-B4-ns和BeiT-base-384 model上进行比较,并提出了一种sparsemax的变体损失函数。该实验使用MSE,MAE,R2,Max MAE/Seed作为主要评价指标(图2),结果显示:BeiT-base-384 model的评价指标明显优于EfficientNet-B4-ns;然后以以BeiT-base-384的结果作为基准,AutoAugment表现最佳;最后确定了最佳的模型为BeiT-base-384 + AutoAug + KLDiv。
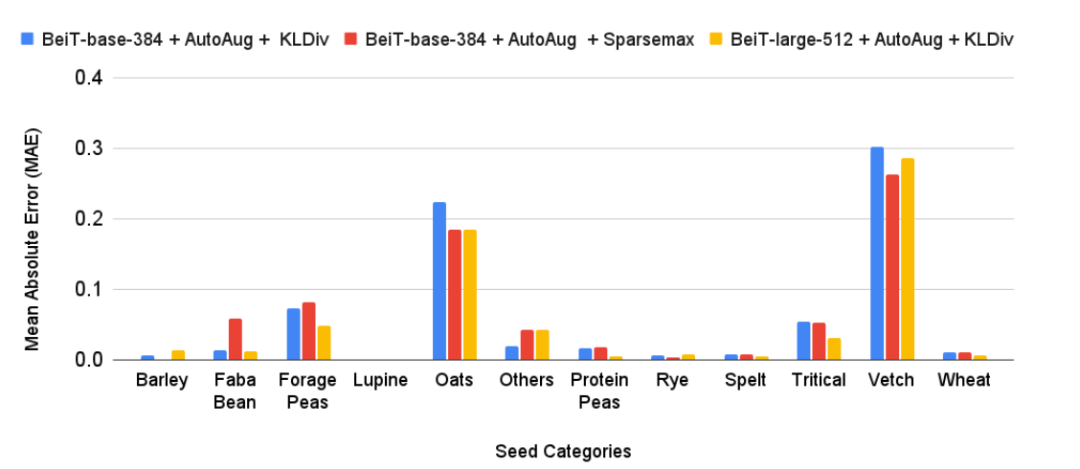
图2使用不同模型架构和损失函数的每种种子类别的MAE比较
从图3可以看出,模型会略微低估一些样品的营养价值,但对大多数样品来说是相当准确的。同时为了评价所选择的模型(BeiT-base-384 + AutoAug + KLDiv)产生的预测营养价值,采用类似的指标来衡量混合种子比例的性能。表1显示了在种子混合物水平预测上取得了改进,其值为0.91。与CNN相比,以自我监督的方式(BeiT)进行预训练的VITS更为准确。性能提高的最可能原因是VIT能够同时捕获全局和局部结构化的内容,从而促进对单个种子的检测和计数。
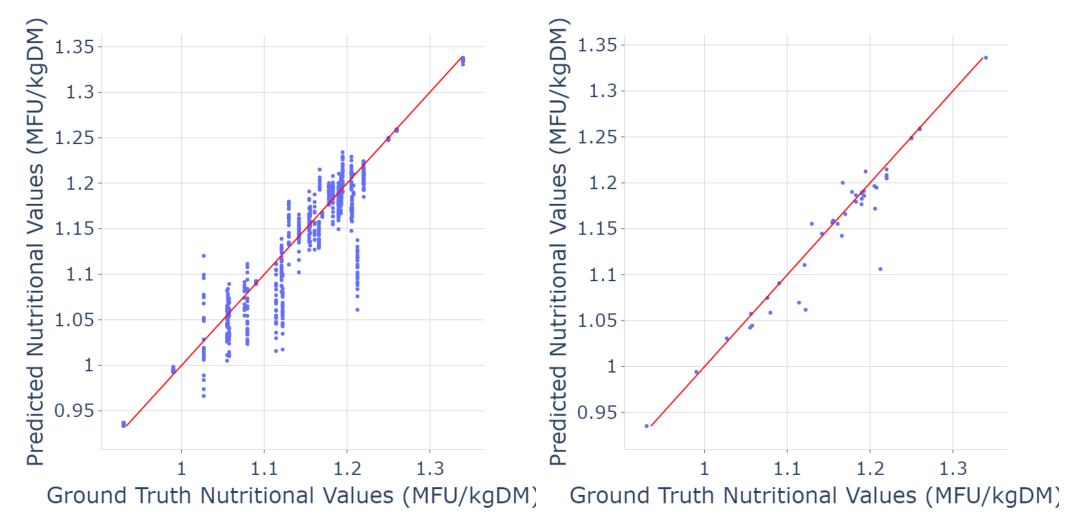
图3图像级营养价值估计(a)和混合级营养价值估计(b)

表1对比图像水平和混合水平的营养价值预测的验证集结果
最终,本研究开发了一个名为"ESTI' METEIL"的网络组件,通过用户界面(https://c4c.inria.fr/carpeso/)对最佳模型进行了试验。该组件允许用户提交1至5张具体种子组合的图像,并获得相应的估计种子组成(按重量)和营养价值。
论文链接:
https://spj.science.org/doi/10.34133/plantphenomics.0112
——推荐阅读——
Improved Field-Based Soybean Seed Counting and Localization with Feature Level Considered
https://doi.org/10.34133/ plantphenomics.0026
Plant Phenomics | 考虑特征水平的田间大豆种子计数和定位的改进
Estimation of Rice Aboveground Biomass by UAV Imagery with Photosynthetic Accumulation Models
https://spj.science.org/doi/10.34133/plantphenomics.0056
Plant Phenomics | 使用无人机图像和光合积累模型估算水稻地上生物量
加入作者交流群
扫码添加小编微信,拉您进入《植物表型组学》作者交流群,群内不定期开展作者分享会、专刊发布会等高质量活动。

添加小编微信,备注姓名+单位+PP,加入作者交流群
About Plant Phenomics
《植物表型组学》(Plant Phenomics)是由南京农业大学和美国科学促进会(AAAS)合作创办的英文学术期刊,于2019年1月正式上线发行。采用开放获取形式,刊载植物表型组学交叉学科热点领域具有突破性科研进展的原创性研究论文、综述、数据集和观点。具体范围涵盖高通量表型分析的最新技术,基于图像分析和机器学习的表型分析研究,提取表型信息的新算法,作物栽培、植物育种和农业实践中的表型组学新应用,与植物表型相结合的分子生物学、植物生理学、统计学、作物模型和其他组学研究,表型组学相关的植物生物学等。期刊已被DOAJ、Scopus、PMC、EI和SCIE等数据库收录。科睿唯安JCR2021影响因子为6.5,位于农艺学、植物科学、遥感一区。中科院农艺学、植物科学一区,遥感二区,生物大类一区(TOP期刊)。2020年入选中国科技期刊卓越行动计划高起点新刊项目。
说明:本文由《植物表型组学》编辑部负责组稿。
中文内容仅供参考,一切内容以英文原版为准。
撰稿:靳松
排版:陈新月
审核:孔敏、王平