品质至上,客户至上,您的满意就是我们的目标
技术文章
当前位置: 首页 > 技术文章
Plant Phenomics | SNAP:基于机器学习的全自动大豆根瘤提取算法
发表时间:2021-09-01 13:28:28点击:1319
来源:植物表型组学
分享:


大豆植株和根瘤菌存在着互惠互利的动态共生关系:在根瘤中,根瘤菌将大气中的氮(N2)固定为植株可利用的氨(NH3),满足了植株对氮元素的需求;作为宿主的大豆植株则会向根瘤菌提供碳元素。氮元素(N)对植株的氨基酸构建、营养生长以及种子内的蛋白质积累至关重要。
近日,Plant Phenomics 在线发表了题为Using Machine Learning to Develop a Fully Automated Soybean Nodule Acquisition Pipeline (SNAP)的研究论文。
大豆根部的根瘤数量从每株几个到几百个不等,在不同基因型的植株之间,根瘤的数量可能有所不同,并沿植株的主根和次生根波动(Figure 1)。在结瘤和未结瘤亲本的比较试验中,生长阶段后期时,结瘤植株中的氮含量能够增加高达6倍,证明了结瘤能够给植株带来积极影响;然而,植株上的根瘤也并非越多越好,目前已有研究证明了高结瘤突变体的效率很低,会导致生物量和产量减少。
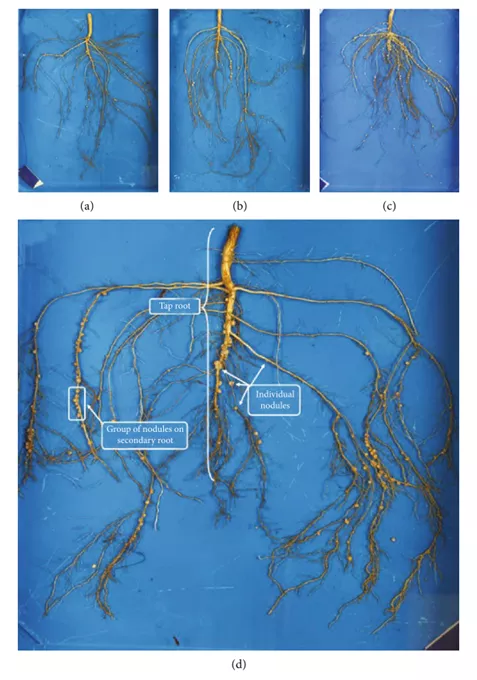
Figure 1: Various genotypes grown in the same field environment with (a) low, (b) moderate, and (c) high nodulation.
对大豆根瘤的定量分析是一项乏味且枯燥的工作。因此,对根瘤的评估通常会在能够进行快速表型分析的尺度上进行,使得信息量较少且具有一定的主观性。
在该文中,作者结合RetinaNet和UNet深度学习架构,提出了用于采集并量化分析大豆根瘤的算法(SNAP,Figure 2),用于对根瘤的检测和分割。该算法使用了来自不同基因型、生长期和田间位置的691个大豆根部数据进行开发,具有良好的模型拟合度(R2=0.99)。该算法降低了人工劳动的强度及其带来的数据偏差,并同时获得与根瘤生长、位置和根分布相关的可量化性状(Figure 3)。由于该算法能够以更高的通量对大豆根部的根瘤进行表型分析,使得研究者能够在生育期早期就对基因和环境因素以及二者的互作对根瘤形成的影响进行评估,从而有望增进研究者对植株与根瘤菌关系的了解。
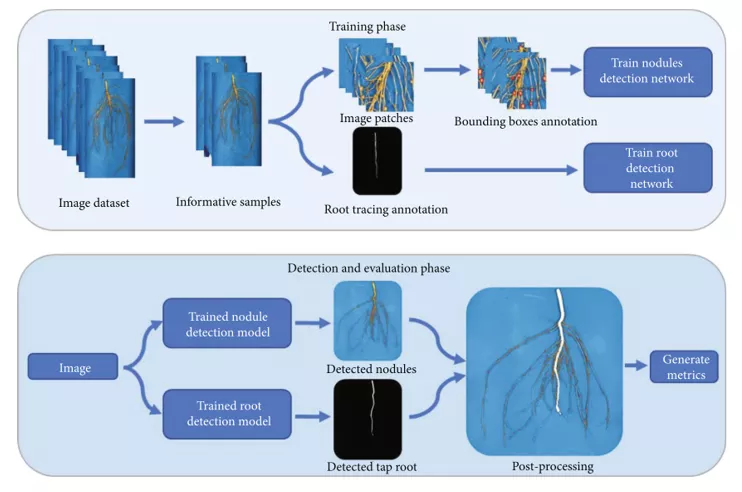
Figure 2:Overview of the workflow of Soybean Nodulation Acquisition Pipeline (SNAP).

Figure 3:(a) Sample input image. (b) SNAP output image with bounding boxes around predicted nodules. (c) SNAP output image with detected taproot (white) and locations of the center of the bounding boxes (red points).
https://spj.sciencemag.org/journals/plantphenomics/2021/9834746/
——推荐阅读——
Qualification of Soybean Responses to Flooding Stress Using UAV-Based Imagery and Deep Learning
https://spj.sciencemag.org/journals/plantphenomics/2021/9892570/
Plant Phenomics | 基于无人机图像和深度学习鉴定大豆对水涝胁迫的响应
Soybean Root System Architecture Trait Study through Genotypic, Phenotypic, and Shape-Based Clusters
https://spj.sciencemag.org/journals/plantphenomics/2020/1925495/
Plant Phenomics | 通过对大豆根系的基因、表型及根形进行聚类分析来研究根系构型特征
《植物表型组学》(Plant Phenomics)是由南京农业大学和美国科学促进会(AAAS)合作创办的英文学术期刊,于2019年1月正式上线发行,是Science合作出版的第二本期刊。采用开放获取形式,刊载植物表型组学交叉学科热点领域具有突破性科研进展的原创性研究论文、综述、数据集和观点。具体范围涵盖高通量表型分析的最新技术,基于图像分析和机器学习的表型分析研究,提取表型信息的新算法,作物栽培、植物育种和农业实践中的表型组学新应用,与植物表型相结合的分子生物学、植物生理学、统计学、作物模型和其他组学研究,表型组学相关的植物生物学等。期刊已被CABI、CNKI、DOAJ、PMC、SCIE和Scopus数据库收录。
说明:本文由《植物表型组学》编辑部负责组稿。
中文内容仅供参考,一切内容以英文原版为准。
撰稿:王栋(实习)
编辑:张威(实习)、鞠笑、孔敏
审核:尹欢