品质至上,客户至上,您的满意就是我们的目标
技术文章
当前位置: 首页 > 技术文章
Plantarray功能生理表型系统:通过生理-转录组学功能方法了解蔬菜豆类中的水分保持与保护特性
发表时间:2022-10-31 15:32:02点击:880
来源:北京博普特科技有限公司
分享:
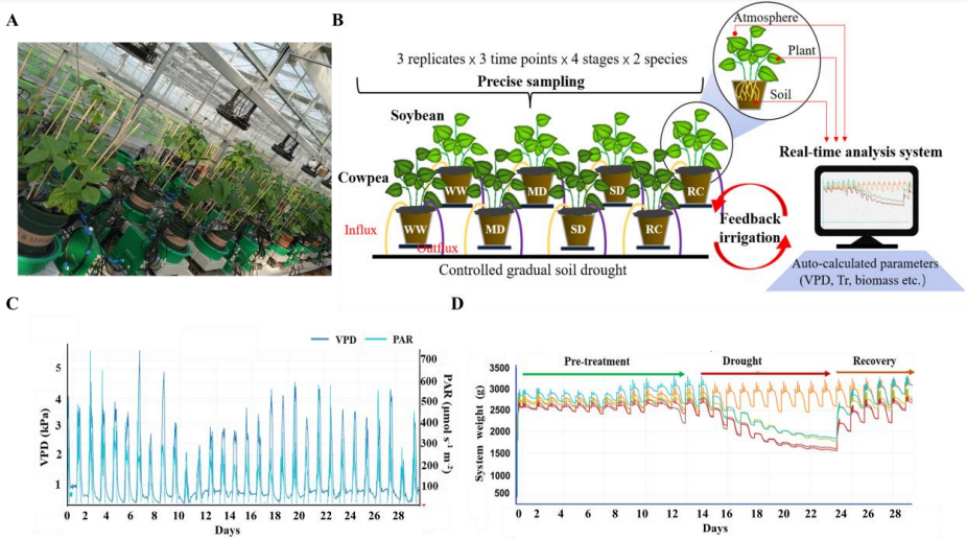
Plantarray是一款基于称重的高通量、多传感器生理表型平台以及植物逆境生物学研究通用平台。该系统可持续、实时测量位于不同环境条件下、阵列中每个植株的土壤-植物-空气(SPAC)中的即时水流动。直接测量根系和茎叶系统水平衡和生物量增加,计算植物生理参数以及植物对动态环境的反馈。系统以有效、易用、无损的方式针对植物对不同处理的反应、预测植物生长和生产力进行定量比较,广泛应用于生物胁迫和非生物胁迫以及植物栽培加速育种研究等,胁迫研究涵盖干旱胁迫、盐胁迫、重金属胁迫、热、冷胁迫、光胁迫以及灌溉/养分、CO2指示、植物健康等领域的研究。

摘要
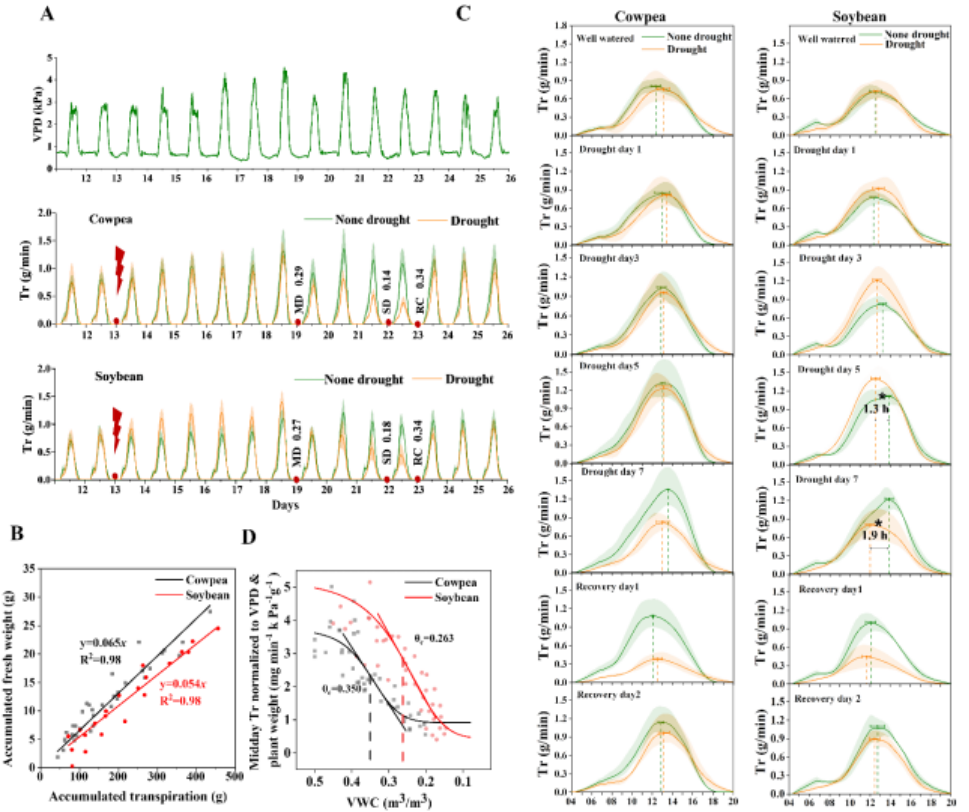
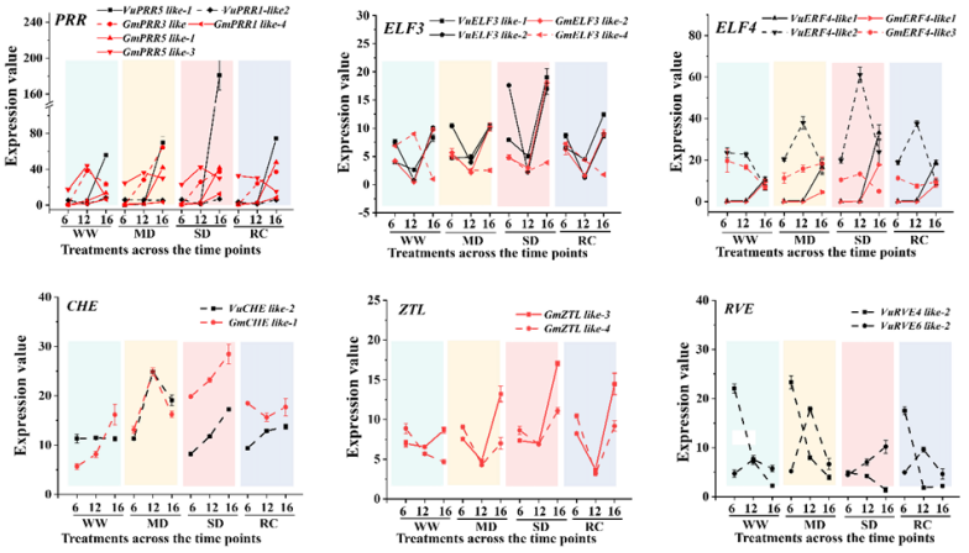
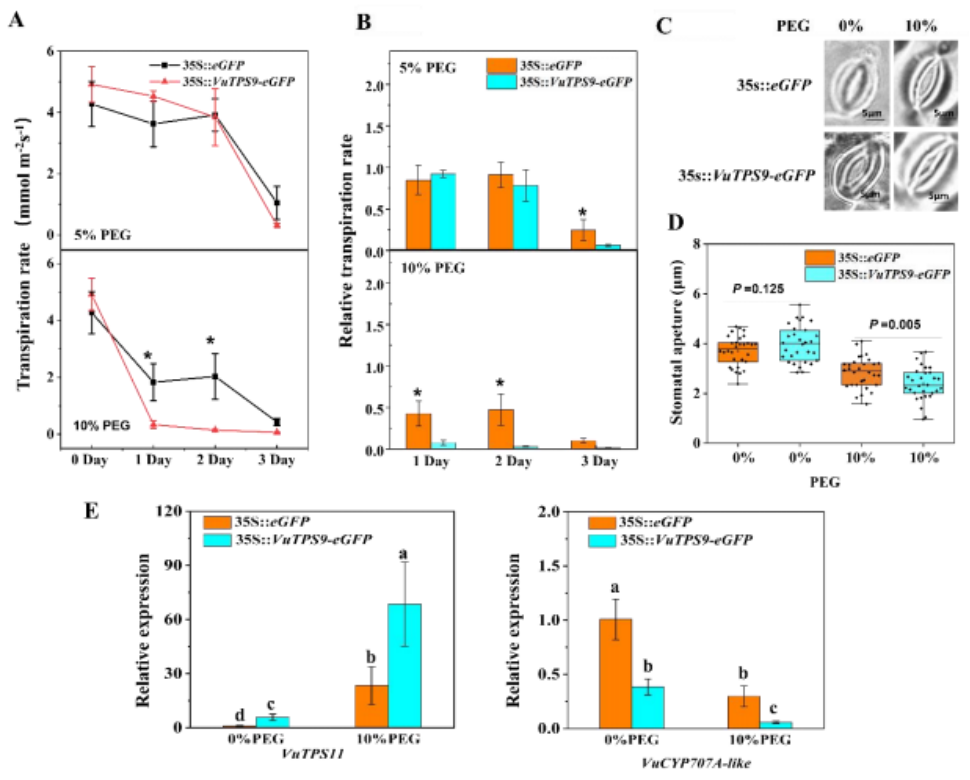
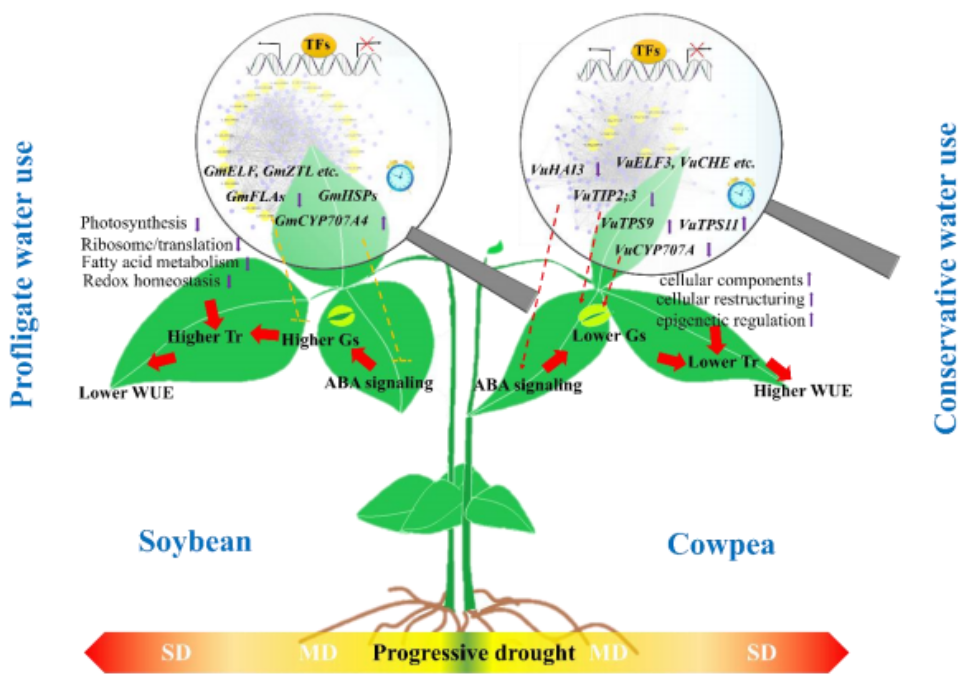
蔬菜大豆和豇豆是相关的暖季豆类,在类似的根系干旱胁迫下叶片水分利用行为表现出鲜明的对比,其机制尚不清楚。在这里,我们对在能够精确控制土壤水分含量的反馈灌溉系统中生长的两种作物进行了综合表型转录组学研究。持续蒸腾速率监测表明,豇豆在早期土壤干旱阶段更保守地使用水分,但在长期干旱下往往保持较高的蒸腾速率。有趣的是,在中度土壤干旱条件下,我们观察到大豆特定蒸腾速率的增加伴随着相移。时间序列转录组学分析表明豇豆在土壤干旱早期的脱水避免机制,其中VuHAI3和VuTIP2;3基因参与。多因素基因聚类分析揭示了基因对干旱、一天中的不同时间以及两种作物之间的相互作用的不同反应,这涉及到核心时钟基因的物种依赖性调节。基因网络分析确定了两个共表达模块,每个模块与豇豆和大豆的蒸腾速率相关,包括物种间的一对负相关模块。鉴定了模块中枢基因,包括ABA降解基因GmCYP707A4和海藻糖磷酸酶/合成酶基因VuTPS9。模块间网络分析揭示了中枢基因的推测的共同参与者。转基因分析证实了VuTPS9在渗透胁迫下调节蒸腾速率的作用。这些发现表明,遭受类似土壤干旱的两种作物叶片中的物种特异性转录组重组不仅是不同抗旱水平的结果,也是其原因之一。
关键词
豇豆;现象学;蔬菜大豆;蒸腾速率;海藻糖磷酸酶/合成酶;水资源关系
Understanding water conservation vs. profligation traits in vegetable legumes through a physio-transcriptomic-functional approach
Abstract
Vegetable soybean and cowpea are related warm-season legumes showing contrasting
leaf water use behaviors under similar root drought stresses, whose mechanisms are not well understood. Here we conducted an integrative phenomic-transcriptomic study on the two crops grown in a feedback irrigation system that enabled precise control of soil water contents. Continuous transpiration rate monitoring demonstrated that cowpea used water more conservatively under earlier soil drought stages, but tended to maintain higher transpiration under prolonged drought. Interestingly, we observed a soybean-specific transpiration rate increase accompanied by phase shift under moderate soil drought. Time -series transcriptomic analysis suggested a dehydration avoidance mechanism of cowpea at early soil drought stage, in which the VuHAI3 and VuTIP2;3 genes were suggested to be involved. Multifactorial gene clustering analysis revealed different responsiveness of genes to drought, time of day and their interactions between the two crops, which involved species -dependent regulation of the core clock genes. Gene network analysis identified two co-expression modules each associated with transpiration rate in cowpea and soybean, including a pair of negatively-correlated modules between species. Module hub genes, including the ABA-degrading gene GmCYP707A4 and the trehalose-phosphatase/synthase gene VuTPS9 were identified. Inter-modular network analysis revealed putative co-players of the hub genes. Transgenic analyses verified the role of VuTPS9 in regulating transpiration rate under osmotic stresses. These findings propose that species-specific transcriptomic reprograming in leaves of the two crops suffering similar soil drought was not only a result of the different drought resistance level, but a cause of it.
Key words
Cowpea; Phenomics; Vegetable soybean; Transpiration rate; Trehalose-phosphatase/synthase; Water relations
相关阅读
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台-烟草研究
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台--番茄研究
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台--番茄研究2
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台--番茄研究3
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台--番茄研究4
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台--番茄研究5
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台-西红柿研究6
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台-拟南芥研究1
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台--拟南芥研究2
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台林木研究-黑松研究
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台林木研究-杨树研究
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台林木研究-柑橘研究
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台-辣椒研究2
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台-辣椒研究3
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台-辣椒研究4
Plantarray高通量植物生理表型平台和植物逆境生物学生理研究平台作物研究-大麦研究2